Il nexiste pas de souris C57BL / 6!
Il est essentiel que vous sachiez quel sous-groupe C57BL / 6 spécifique vous utilisez afin dutiliser les contrôles appropriés pour vos expériences et dinterpréter correctement vos données! Depuis C.C. Little (le fondateur du Jackson Laboratory) a initialement généré la souche consanguine C57BL dans les années 1920-1930, la souche consanguine C57BL / 6 est devenue la souche de souris la plus fréquemment utilisée dans la recherche biomédicale. La popularité des souris consanguines C57BL / 6 a conduit à létablissement de nombreuses colonies chez différents fournisseurs et établissements universitaires à travers le monde.
Sous-souches C57BL / 6
Vous ne le savez peut-être pas: chaque Lorsquune nouvelle colonie C57BL / 6 est maintenue séparément dune colonie existante pendant 20 générations ou plus, elle devient un nouveau sous-groupe C57BL / 6. Les générations sont cumulatives, donc si chacune des deux colonies distinctes se reproduit pendant 10 générations (~ 2-3 ans), elles sont séparées de 20 générations et différentes sous-souches avec des phénotypes potentiellement différents. Dans le cadre de la nomenclature d’une souche, les codes de laboratoire sont ajoutés à la fin en tant que désignation de sous-groupe. C57BL / 6J est le sous-groupe parental; « J » est le code de laboratoire du Jackson Laboratory. Par conséquent, il ny a pas de source de souris « C57BL / 6 »; il y a toujours une désignation plus longue pour chaque sous-groupe indiquant linstitut ou le laboratoire qui maintient les différentes colonies.
Les sous-souches C57BL / 6 ne sont pas les mêmes!
Une fois une nouvelle sous-formation C57BL / 6 est établie, des mutations spontanées surviendront à la fois dans la colonie dorigine et dans la nouvelle colonie. Un sous-ensemble de ces mutations se propage à travers la colonie par dérive génétique et se fixe (homozygote chez toutes les souris). Plus les sous-souches individuelles sont séparées les unes des autres, plus il y a de différences génétiques entre elles. Ces différences génétiques peuvent conduire à des différences phénotypiques.
C57BL / 6J vs C57BL / 6N
En 1951, des souris C57BL / 6J ont été envoyées aux National Institutes of Health (NIH) où une colonie a été établie et a été appelée C57BL / 6N. Par la suite, de nombreux sous-souches ont été dérivés de la colonie C57BL / 6N. Une mutation qui provoque une dégénérescence rétinienne inégale, connue sous le nom de Crb1rd8, a été découverte comme étant homozygote dans toutes les sous-souches liées à C57BL / 6N, mais nest pas présente dans la sous-séquence C57BL / 6J. De plus, les données recueillies auprès des centres de phénotypage IKMC (International Knockout Mouse Consortium) ont révélé de nombreuses différences phénotypiques entre les sous-souches C57BL / 6J et C57BL / 6N.
Les dangers de tomber dans le piège de lignorance
Il peut y avoir des conséquences graves si vous nêtes pas pleinement conscient du fond génétique (souche et sous-groupe) ou de vos souris expérimentales. Vous ne seriez pas le premier chercheur à tomber dans ce piège. Lorsque vous choisissez la mauvaise souche de contrôle, vous avez un risque élevé de mal interpréter vos données, de tirer des conclusions erronées et de retarder sérieusement votre programme de recherche.
Notre article de blog, « Pourquoi il a fallu 2 ans à un laboratoire de recherche de Harvard pour revenir à la recherche » décrit comment un laboratoire de recherche a accidentellement associé un phénotype dimmunodéficience à un allèle knock-out alors quen fait cétait dû à une mutation du sous-groupe C57BL / 6 particulier sur lequel le knockout a été rétrocroisé. Lerreur a été découverte lorsque le modèle knockout a été rétrocroisé avec un sous-groupe C57BL / 6 dun autre fournisseur, et le phénotype a été perdu. La plupart du temps, effort, et les ressources utilisées pour élucider pourquoi léquipe na pas pu reproduire les résultats précédents auraient pu être sauvegardées si les auteurs avaient utilisé des souris témoins avec le même bagage génétique – que celui utilisé pour rétrocroiser le modèle KO dintérêt.
Ce gène est-il protecteur ou toxique?
Un autre exemple notable provient dun laboratoire du National Heart, Lung, and Blood Institute (qui fait partie du NIH). Après avoir testé les effets dun knock-out Mapk9 (Jnk2) sur lacétaminophène – lésion hépatique induite en utilisant C57BL / 6J comme témoins de type sauvage , les résultats étaient contraires aux attentes. Lorsquil est répété en utilisant C57BL / 6NJ (C57BL / 6N importé dans JAX du NIH en 2005) comme témoins de type sauvage, le phénotype des knockouts Mapk9 se situe carrément entre le phénotype du C57BL / 6J et du C57BL / 6NJ (voir figure).
Les chercheurs se sont retrouvés dans une situation où ils pouvaient interpréter leurs données de deux manières opposées, selon le contrôle utilisé. Si le C57BL / 6J utilisé comme contrôle, les données indiquent que MAPk9 est hépatoprotecteur. Sils utilisaient C57BL / 6NJ comme contrôles, alors MAPK9 semblait être hépatotoxique.
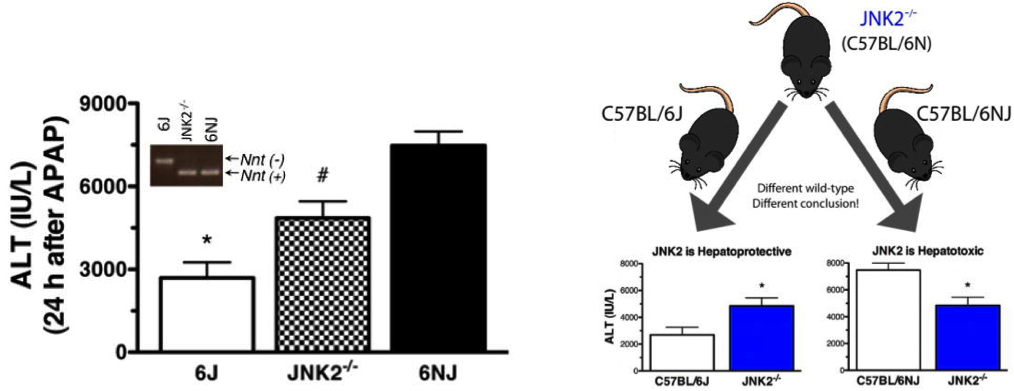
Figure 1. Conclusions sur les données diffèrent selon la sélection de la souche de contrôle. Les souris ont été traitées avec de lacétaminophène (APAP, 300 mg / kg par voie intrapéritonéale). Les lésions hépatiques ont été évaluées 24 heures après le traitement en mesurant lactivité sérique de lalanine aminotransférase (ALT).
Heureusement, les chercheurs ont pu déterminer que le knock-out de Mapk9 était sur un fond C57BL / 6N, et ont conclu que le gène était hépatotoxique.Cependant, pensez à la facilité avec laquelle ces données ont pu être mal interprétées et à la fréquence à laquelle de telles erreurs sont complètement manquées, conduisant à des résultats non reproductibles!
Par conséquent, soyez prévenu et assurez-vous que vous connaissez non seulement la souche consanguine, mais aussi le sous-groupe de souris que vous utilisez pour les expériences afin de choisir les bons contrôles et de produire des données fiables et significatives. Souvenez-vous quil ny a pas de souche de souris C57BL / 6, il y a toujours un nom plus long!